Študijske metode, diagnostika, bolezni, bolezni
- 2060
- 84
- Raymond Moen
A Haplootype To je območje genoma, ki se ponavadi podeduje skozi več generacij; Običajno se vse nahaja v istem kromosomu. Haplootipi so produkt genetske vezi in ostanejo nedotaknjeni med genetsko rekombinacijo.
Beseda "haplotipo" izhaja iz kombinacije besede "haploid" in besede "genotip". "Haploid" se nanaša na celice z enim naborom kromosomov in "genotip" govori o genetski sestavi organizma.
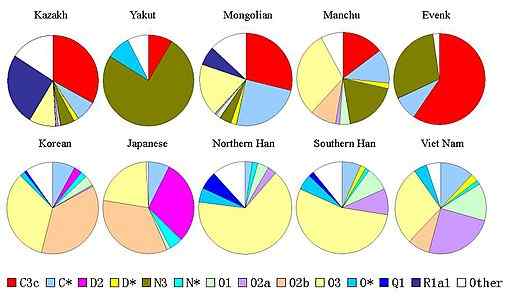
 Shema porazdelitve kromosomskih haplotipov in v azijskih populacijah (vir: Moogalord [javna domena] prek Wikimedia Commons) Po definiciji lahko haplotip opiše nekaj genov ali več, ki so podedovani na kromosomu iz staršev lahko opiše kromosom, ki je popolnoma podedovan iz staršev, prav tako primer kromosoma in moških.
Shema porazdelitve kromosomskih haplotipov in v azijskih populacijah (vir: Moogalord [javna domena] prek Wikimedia Commons) Po definiciji lahko haplotip opiše nekaj genov ali več, ki so podedovani na kromosomu iz staršev lahko opiše kromosom, ki je popolnoma podedovan iz staršev, prav tako primer kromosoma in moških.
Na primer, ko haplotipi delijo gene za dva različna fenotipska lika, kot sta barva las in barva oči, bodo posamezniki, ki imajo gen za barvo, imeli tudi drugi gen za barvo oči.
Haplootipi so danes eno najbolj uporabljenih orodij za preučevanje rodoslovja, za sledenje izvoru bolezni, za karakterizacijo genetske variabilnosti in filogeografije populacij različnih vrst živih bitij.
Obstaja več orodij za preučevanje halootipov, eno najbolj uporabljenih je danes. "Zemljevid haplotipa»(Hapmap), ki je spletna stran, ki omogoča določitev segmentov genoma, ki so haplotipi.
[TOC]
Metode študija
Haplootipi predstavljajo priložnost za razumevanje dedovanja genov in njihovega polimorfizma. Z odkritjem tehnike "verižna reakcija polimeraze" (PCR, angleščina "Verižna reakcija polimeraze”) Je široko napredoval pri preučevanju haplotipov.
Trenutno obstajajo številne metodologije za preučevanje haplotipov, nekateri največ vrhuncev so:
Zaporedje DNK in odkrivanje enojnega nukleotidnega polimorfizma (SNP)
Razvoj tehnologij zaporedja nove generacije je predstavljal velik preskok za preučevanje haplotipov. Nove tehnologije omogočajo razlike do ene same nukleotidne baze v določenih regijah haplotipa.
Vam lahko služi: rekombinantna DNK: tehnika, aplikacije in temeljeV bioinformatiki se izraz haplotip uporablja tudi za navajanje dedovanja enega samega nukleotidnega polimorfizma (SNP) v sekvencah DNK.
Združevanje bioinformatičnih programov z odkrivanjem haplotipov z uporabo zaporedja nove generacije je mogoče natančno prepoznati položaj, zamenjavo in učinek sprememb vsake baze na genom populacije.
Mikrosateliti (SSR)
Mikrosateliti ali SSR izhajajo iz angleščine "Simplementirati ponovitev in Kratka ponovitev tandema". To so kratka nukleotidna sekvence, ki se v genomskem območju ponavljajo zaporedno.
Običajno je najti mikrosatelite znotraj nekodirajočih haplotipov, zato je z odkrivanjem variacij števila ponovitev mikrosatelitov mogoče opaziti različne alele v haplotipih posameznikov.
Mikrosatelitski molekularni markerji so bili razviti za odkrivanje neskončnih haplotipov, iz seksata rastlin, kot je papaja (Carica papaya) do odkrivanja človeških bolezni, kot je falciformna anemija.
Polimorfizmi amplificiranih fragmentov (AFLP)
Ta tehnika združuje amplifikacijo z reakcijami PCR z prebavo DNK z dvema različnima restrikcijskima encimima. Tehnika zazna polimorfne lokuse v haplootipih glede na različna mesta rezanja v zaporedju DNK.
Če želite bolje prikazati tehniko, si predstavljajmo tri fragmente tkanine iste dolžine, vendar razrežemo na različne kraje (ti fragmenti predstavljajo tri fragmente amplificiranih haplotipov s pomočjo PCR tehnike).
V času, ko je tkanina razrezana, bo doseženih veliko kosov različnih velikosti, saj se vsaka krpa razreže na različne kraje. Pri naročanju fragmentov glede na vrsto tkanine, iz katere prihajajo, lahko opazimo, kje najdemo razlike med tkaninami ali haplotipi.
Diagnostika in bolezni
Pomembna prednost genetske študije haplotipov je, da ti na tisoče generacij ostanejo skoraj nepoškodovani ali nespremenjeni, kar omogoča identifikacijo oddaljenih prednikov in vsake mutacije, s katerimi posamezniki prispevajo k razvoju bolezni.
Vam lahko služi: fenotipske različiceHaplootipi v človeštvu se razlikujejo glede na rase in od te zajemalke so bili v haplotipih odkriti geni, ki povzročajo hude bolezni pri vsaki od človeških ras.
V projektu Hapmap Vključene so štiri rasne skupine: evropska, nigerijska Yoruba, kitajski Han in japonščina.
Na ta način projekt Hapmap Lahko pokrije različne skupine populacij in sledi izvoru in evoluciji številnih dednih bolezni, ki vplivajo na vsako od štirih dirk.
Ena od bolezni, ki se najpogosteje diagnosticira z analizo haplotipa, je falciformna anemija pri ljudeh. Ta bolezen se diagnosticira s sledenjem pogostosti afriških haplotipov v populacijo.
Ker je bolezen, ki izvira iz Afrike, prepoznavanje afriških haplotipov v populaciji omogoča, da zlahka sledite ljudem, ki imajo mutacijo v genetskem zaporedju beta globunine eritrocitov v obliki HOZ (značilna za patologijo).
Primeri
S haplotipi filogenetska drevesa, ki predstavljajo evolucijska razmerja med vsakim haplotipom, ki jih najdemo v vzorcu homolognih molekul DNK ali istih vrst, v regiji, ki ima malo ali nič rekombinacije, se gradijo.
Ena najbolj preučenih vej s haplotipi je razvoj imunskega sistema ljudi. Haplootipi, ki koda za receptor za cestnino (ključna sestavina prirojenega imunskega sistema) so bili ugotovljeni za genom Neandertal in Denisovanos.
To jim omogoča, da izsledijo, kako so se genetske sekvence spremenile v populacijah "modernih" ljudi glede na haplotipne sekvence, ki ustrezajo "ljudem prednikov".
Gradnja mreže genetskih odnosov iz mitohondrijskega haplotipa.
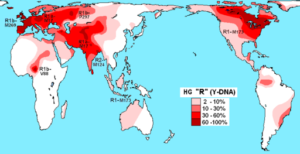 Porazdelitev haplogrupa R (DNA-Y) pri domačih populacijah (vir: Maucioni [CC do 3.0 (https: // creativeCommons.Org/licence/by/3.0)], prek Wikimedia Commons) se raznolikost haplotipov uporablja za sledenje in preučevanje genetske raznolikosti živali, vzgojenih v ujetništvu. Te tehnike se uporabljajo posebej za težke vrste za spremljanje prostoživečih živali.
Porazdelitev haplogrupa R (DNA-Y) pri domačih populacijah (vir: Maucioni [CC do 3.0 (https: // creativeCommons.Org/licence/by/3.0)], prek Wikimedia Commons) se raznolikost haplotipov uporablja za sledenje in preučevanje genetske raznolikosti živali, vzgojenih v ujetništvu. Te tehnike se uporabljajo posebej za težke vrste za spremljanje prostoživečih živali.
Živalske vrste, kot so morski psi, ptice in veliki sesalci, kot so jaguarji, sloni, med drugim nenehno gensko ocenjujejo z mitohondrijskimi haplotipi za spremljanje genetskega stanja populacije v ujetništvu.
Reference
- Bahlo, m., Stankovič, j., Hitrost, t. Str., Rubio, j. Str., Burfoot, r. K., & Stopalo, s. J. (2006). Delitev genoma široke haplotipe z uporabo podatkov SNP ali mikrosatelitskega haplotipa. Človeška genetika, 119 (1-2), 38-50.
- Dannemann, m., Andrés, a. M., & Kelso, J. (2016). Introgres neandertal-indo denisovanov podobnih haplotipov prispeva k prilagodljivi razliki v človeških cestninskih sprejemnikih. Ameriški časopis za človeško genetiko, 98 (1), 22–33.
- Iz Vries, h. G., Van der Meulen, m. Do., Rozen, r., Halley, d. J., Scheffer, h., Leo, str.,... & ti meerman, g. J. (devetnajst devetdeset šest). Haplotipna identiteta med posamezniki, ki imajo alel za mutacijo CFTR. Človeška genetika, 98 (3), 304–309
- Lastovka, m. Do., Oganje, a. L., Christiansen, f. T., Witt, c. S., Abraham, l. J., & Dawkins, r. L. (1992). Predniki prednikov: konzerva populacije MHC haplotipi. Človeška imunologija, 34 (4), 242-252.
- Štipendisti, m. R., Hartman, t., Hermelin, d., Landau, g. M., Rosamond, f., & Rozenberg, L. (2009, junij). Haplotip sklep, ki ga omejujejo verodostojni haplotipni podatki. V letnem simpoziju o ujemanju kombiniranega vzorca (PP. 339-352). Springer, Berlin, Heidelberg.
- Gabriel, s. B., Schaffner, s. F., Nguyen, h., Moore, j. M., Roy, j., Blumenstiel, b.,... & liu-cordero, s. N. (2002). Struktura haplotipa v človeškem genomu. Science, 296 (5576), 2225-2229.
- Mednarodni konzorcij HAPMAP. (2005). Haplotip zemljevid človeškega genoma. Nature, 437 (7063), 1299.
- Wynne, r., & Wilding, c. (2018). Mitohondrijska raznolikost haplotipa DNK in izvor morskih psov v ujetništvu (Carcharias Taurus). Journal of Zoo in Aquarium Research, 6 (3), 74–78.
- Yoo in. J., Tang, j., Kaslow, r. Do., & Zhang, k. (2007). Haplotip sklepa o sedanjih podatkih o genotipu z uporabo predvoje identificiranih haplotipov in haplotipov. Bioinformatics, 23 (18), 2399-2406.
- Mladi, n. S. (2018). Aplastična anemija. New England Journal of Medicine, 379 (17), 1643-1656.
- « Hollandske značilnosti dedovanja, funkcije genov, degeneracija
- Lentén večje značilnosti, habitat, lastnosti, nega »

